از طریق منوی جستجو مطلب مورد نظر خود در وبلاگ را به سرعت پیدا کنید
سیستم AI قطعات پروتئینی را پیش بینی می کند که می توانند به یک هدف متصل شوند یا مهار کنند
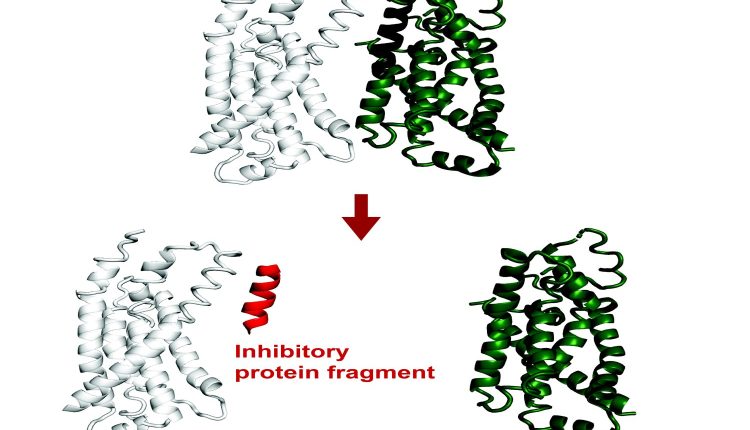
تمام عملکرد بیولوژیکی وابسته است روی چگونه پروتئین های مختلف با یکدیگر تعامل دارند. فعل و انفعالات پروتئین پروتئین همه چیز را از رونویسی DNA و کنترل تقسیم سلولی به عملکردهای سطح بالاتر در ارگانیسم های پیچیده تسهیل می کند.
با این حال ، در مورد چگونگی ارکستر این توابع بسیار نامشخص است روی سطح مولکولی ، و چگونگی تعامل پروتئین ها با یکدیگر – چه با پروتئین های دیگر و چه با نسخه های خود.
یافته های اخیر نشان داده است که قطعات پروتئین کوچک پتانسیل عملکردی زیادی دارند. حتی اگر آنها قطعات ناقص هستند ، اما کشش های کوتاه اسیدهای آمینه هنوز هم می توانند به رابط های یک پروتئین هدف متصل شوند ، و تعامل بومی را مجدداً مورد استفاده قرار می دهند. از طریق این processآنها می توانند عملکرد پروتئین را تغییر دهند یا تعامل آن با پروتئین های دیگر را مختل کنند.
بنابراین قطعات پروتئین می توانند هر دو تحقیق اساسی را توانمند سازند روی فعل و انفعالات پروتئین و فرآیندهای سلولی ، و به طور بالقوه می تواند کاربردهای درمانی داشته باشد.
اخیراً منتشر شده در مجموعه مقالات آکادمی ملی علوم، روشی جدید که در گروه زیست شناسی ایجاد شده است روی مدلهای هوش مصنوعی موجود برای پیش بینی محاسباتی قطعات پروتئینی که می توانند پروتئین های تمام طول را در آن متصل و مهار کنند E. coliبشر از لحاظ تئوریکی ، این ابزار می تواند منجر به مهارکننده های قابل رمزگذاری ژنتیکی در برابر هر پروتئین شود.
این کار در آزمایشگاه دانشیار زیست شناسی و محقق موسسه پزشکی هوارد هیوز با همکاری آزمایشگاه جی A. استین (1968) استاد زیست شناسی ، استاد مهندسی بیولوژیکی و رئیس بخش امی کیتینگ انجام شد.
اهرم یادگیری ماشین
این برنامه با نام Fragfold ، Alphafold را به دست می آورد ، یک مدل هوش مصنوعی که به دلیل توانایی آن در پیش بینی تاشو پروتئین و تعامل پروتئین منجر به پیشرفت های پدیده ای در زیست شناسی شده است.
هدف این پروژه پیش بینی مهار کننده های قطعه ای بود که یک کاربرد جدید از Alphafold است. محققان روی این پروژه به صورت تجربی تأیید کرد که بیش از نیمی از پیش بینی های Fragfold برای اتصال یا مهار دقیق بودند ، حتی اگر محققان اطلاعات ساختاری قبلی نداشتند روی مکانیسم آن تعامل.
“نتایج ما نشان می دهد که این یک روش قابل تعمیم برای یافتن حالت های اتصال دهنده است که احتمالاً عملکرد پروتئین را مهار می کنند ، از جمله برای اهداف پروتئین جدید ، و شما می توانید از این پیش بینی ها به عنوان نقطه شروع برای آزمایش های بیشتر استفاده کنید.” اندرو ساوینوف ، یک پست دکتری در آزمایشگاه لی. “ما واقعاً می توانیم این کار را در پروتئین ها بدون توابع شناخته شده ، بدون تعامل شناخته شده ، بدون ساختارهای شناخته شده ، اعمال کنیم و می توانیم در این مدل هایی که در حال توسعه هستیم ، اعتبار خود را ایجاد کنیم.”
یک مثال FTSZ است ، پروتئینی که برای تقسیم سلولی مهم است. این مطالعه به خوبی مورد مطالعه قرار گرفته است اما حاوی منطقه ای است که ذاتاً بی نظم است و بنابراین ، به ویژه برای مطالعه چالش برانگیز است. پروتئین های بی نظم پویا هستند ، و تعامل عملکردی آنها به احتمال زیاد زودگذر است – به طور خلاصه اتفاق می افتد که ابزارهای زیست شناسی ساختاری فعلی نمی توانند یک ساختار یا تعامل واحد را ضبط کنند.
محققان برای کشف فعالیت قطعات FTSZ ، از جمله قطعاتی از منطقه ذاتاً بی نظم ، برای شناسایی چندین تعامل اتصال جدید با پروتئین های مختلف ، از Fragfold استفاده کردند. این جهش در درک آزمایش های قبلی اندازه گیری فعالیت بیولوژیکی FTSZ را تأیید و گسترش می دهد.
این پیشرفت تا حدودی قابل توجه است زیرا بدون حل ساختار منطقه بی نظم انجام شده است ، و به دلیل این که قدرت بالقوه Fragfold را نشان می دهد.
کیتینگ می گوید: “این یک نمونه از چگونگی تغییر در تغییر در روش تغییر چگونگی مطالعه زیست شناسی مولکولی و سلولی است.” “برنامه های خلاقانه از روش های هوش مصنوعی ، مانند کار ما روی شکننده ، قابلیت های غیر منتظره و مسیرهای تحقیق جدید را باز کنید. “
مهار ، و فراتر از آن
محققان این پیش بینی ها را با تکه تکه کردن محاسبات هر پروتئین انجام دادند و سپس الگوبرداری از چگونگی اتصال این قطعات به شرکای تعامل مورد نظر که فکر می کردند مرتبط هستند.
آنها نقشه های اتصال پیش بینی شده را در کل دنباله با اثرات همان قطعات در سلولهای زنده ، با استفاده از اندازه گیری های تجربی با توان بالا که در آن میلیون ها سلول هر یک از یک نوع قطعه پروتئین تولید می کنند ، مقایسه کردند.
Alphafold از اطلاعات همبستگی برای پیش بینی تاشو استفاده می کند ، و به طور معمول تاریخ تکاملی پروتئین ها را با استفاده از چیزی به نام ترازهای توالی چندگانه برای هر پیش بینی انجام می دهد. MSA بسیار مهم است ، اما یک تنگنا برای پیش بینی های در مقیاس بزرگ است-آنها می توانند مدت زمان و قدرت محاسباتی را ممنوع بگیرند.
در عوض ، محققان MSA را برای یک پروتئین تمام طول یک بار محاسبه کردند و از این نتیجه استفاده کردند تا پیش بینی های هر قطعه از آن پروتئین تمام طول را هدایت کنند.
Savinov ، همراه با دانشکده آزمایشگاه Keating Sebastian Swanson PhD ’23 ، علاوه بر FTSZ ، قطعات مهاری از مجموعه متنوعی از پروتئین ها را پیش بینی کرد. در بین فعل و انفعالات مورد بررسی ، پیچیده ای بین پروتئین های حمل و نقل لیپوپلی ساکارید LPTF و LPTG بود. قطعه پروتئینی LPTG این تعامل را مهار می کند ، احتمالاً اختلال در تحویل لیپوپلی ساکارید ، که یک مؤلفه مهم از آن است E. coli غشای سلول بیرونی برای تناسب اندام سلولی ضروری است.
ساوینوف می گوید: “تعجب بزرگ این بود که ما می توانیم با چنین دقت بالایی اتصال را پیش بینی کنیم و در واقع ، اغلب اتصال را که مربوط به مهار است پیش بینی می کند.” “برای هر پروتئینی که به آن نگاه کرده ایم ، توانسته ایم مهار کننده ها را پیدا کنیم.”
محققان در ابتدا متمرکز شدند روی قطعات پروتئینی به عنوان مهارکننده ها به این دلیل که آیا یک قطعه می تواند یک عملکرد اساسی در سلول ها را مسدود کند ، یک نتیجه نسبتاً ساده برای اندازه گیری سیستماتیک است. با نگاهی به جلو ، Savinov همچنین علاقه مند به کاوش در عملکرد قطعه در خارج از مهار ، مانند قطعاتی است که می توانند پروتئینی را که به آنها متصل می شوند ، تثبیت کنند ، عملکرد آن را تقویت یا تغییر دهند یا باعث تخریب پروتئین شوند.
طراحی ، در اصل
این تحقیق یک نقطه شروع برای ایجاد درک سیستمیک از اصول طراحی سلولی است ، و چه عناصر مدل های یادگیری عمیق ممکن است ترسیم کنند روی برای پیش بینی های دقیق.
ساوینوف می گوید: “یک هدف گسترده تر و بیشتر با دست و پاگیر وجود دارد که ما در حال ساختن آن هستیم.” “اکنون که می توانیم آنها را پیش بینی کنیم ، آیا می توانیم از داده های مورد نظر خود از پیش بینی ها و آزمایشات استفاده کنیم تا ویژگی های برجسته را بیرون بیاوریم تا بفهمیم چه Alphafold در واقع در مورد آنچه که باعث ایجاد یک مهار کننده خوب شده است؟”
Savinov و همکاران همچنین در مورد چگونگی اتصال قطعات پروتئین ، به بررسی سایر فعل و انفعالات پروتئین و جهش باقیمانده های خاص برای دیدن اینکه چگونه این تعامل ها روش تعامل این قطعه با هدف خود تغییر می دهند ، بیشتر می پردازند.
به طور تجربی با بررسی رفتار هزاران قطعه جهش یافته در سلول ها ، رویکردی که به عنوان اسکن جهش یافته عمیق شناخته می شود ، اسیدهای آمینه کلیدی را نشان داد که مسئول مهار هستند. در بعضی موارد ، قطعات جهش یافته حتی مهار کننده های قوی تر از توالی های طبیعی و تمام قد آنها بودند.
سوانسون می گوید: “برخلاف روشهای قبلی ، ما محدود به شناسایی قطعات در داده های ساختاری تجربی نیستیم.” “قدرت اصلی این کار تعامل بین داده های مهار تجربی با توان بالا و مدلهای ساختاری پیش بینی شده است: داده های تجربی ما را به سمت قطعاتی که به ویژه جالب هستند راهنمایی می کند ، در حالی که مدل های ساختاری پیش بینی شده توسط Fragfold یک فرضیه خاص و قابل آزمایش را برای شما فراهم می کنند چگونه قطعات عملکرد دارند روی یک سطح مولکولی. “
Savinov از آینده این رویکرد و برنامه های بی شمار آن هیجان زده است.
لی موافق است: “با ایجاد اتصال دهنده های جمع و جور ، از نظر ژنتیکی ، Fragfold طیف گسترده ای از امکانات را برای دستکاری عملکرد پروتئین باز می کند.” “ما می توانیم تصور کنیم که قطعات کاربردی را ارائه می دهیم که می توانند پروتئین های بومی را تغییر دهند ، محلی سازی درون سلولی آنها را تغییر دهند و حتی آنها را مجدداً برنامه ریزی کنند تا ابزارهای جدیدی برای مطالعه زیست شناسی سلولی و درمان بیماری ها ایجاد کنند.”
منبع: https://news.mit.edu/1404/ai-system-fragfold-predicts-protein-fragments-0220
لطفا در صورت وجود مشکل در متن یا مفهوم نبودن توضیحات، از طریق دکمه گزارش نوشتار یا درج نظر روی این مطلب ما را از جزییات مشکل مشاهده شده مطلع کنید تا به آن رسیدگی کنیم
زمان انتشار: 1404-02-21 11:04:09



